„Alles wurscht?!“ – Tierartendifferenzierung mittels Polymerasekettenreaktion
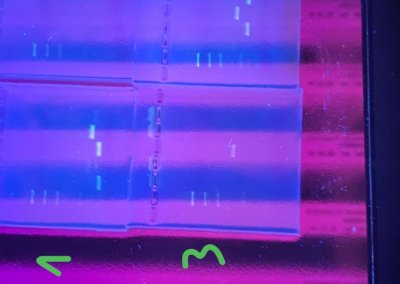
Unser Biologieleistungskurs der Q1 besuchte gemeinsam mit Frau Gocke in Form einer Tagesexkursion das Biolabor b!lab in Beverungen. Im Rahmen der aktuellen Unterrichtsinhalte in diesem Schulhalbjahr konnten wir unsere bisherigen Kenntnisse aus dem Bereich der Genetik anwenden und vertiefen. Erstmalig gewannen wir einen Einblick in das Forschungsgebiet der Gentechnik, das als ausgewiesener Abiturschwerpunkt in den kommenden Wochen weiterhin Unterrichtsgegenstand sein wird. In unserem Kurs behandelten wir den Sachverhalt der Verunreinigung von Fleischprodukten. Wir untersuchten Hähnchenprodukte auf Beimengungen von Putenfleisch sowie Rinderprodukte auf Schweinefleisch, indem wir das Erbgut, die Desoxyribonucleinsäure (DNA), aus Zellen der jeweiligen Tierprodukte isolierten, mittels der Polymerasekettenreaktion (PCR) vervielfältigten und das Ergebnis mit einer Gelelektrophorese nachwiesen. Für die PCR kamen tierartenspezifische Primer zum Einsatz. Diese Primer sind kurze RNA-Fragmente (RNA = Ribonucleinsäure) und ermöglichen die Vervielfältigung bestimmter DNA-Fragmente, sofern sie in einer Probe vorhanden sind. Die Gelelektrophorese trennt die vervielfältigten DNA-Fragmente im Anschluss nach ihrer Größe auf. Das dabei entstehende Bandenmuster kann mit einem Marker bekannter Fragmente verglichen werden. Anhand des Vergleichs kann dann auf das Vorkommen des Erbguts bestimmter Tierarten geschlossen werden. Der Besuch des b!lab ermöglichte uns einen praktischen Einblick in die Laborarbeit sowie das Forschungsgebiet der Gentechnik. Mit den gewonnenen Erkenntnissen blicken wir motiviert und interessiert auf die Unterrichtsstunden nach den Osterferien, in denen wir die Gentechnik vertiefen und weitere Methoden kennenlernen.